Chapitre 2 Les méthodes MCMC
2.1 Introduction
J’espère que je ne vous ai pas (trop) perdu dans le chapitre précédent avec toutes ces équations. Dans ce nouveau chapitre, nous passons dans les coulisses de la statistique bayésienne, en découvrant les méthodes de Monte Carlo par chaînes de Markov (MCMC). Vous verrez comment et pourquoi ces techniques de simulation sont devenues essentielles pour mettre en œuvre l’inférence bayésienne en pratique. Et comme rien ne vaut la pratique, nous mettrons un peu la main à la pâte en codant nous-mêmes, à travers notre exemple fil rouge sur l’estimation d’une probabilité de survie.
2.2 Application du théorème de Bayes
Revenons à notre exemple fil rouge sur les ragondins, je redonne les données :
y <- 19 # nombre d'individus ayant survécu à l'hiver
n <- 57 # nombre d'individus suivis au début de l'hiverAppliquons le théorème de Bayes de manière plus directe que dans le Chapitre 1, dans lequel on a mis de côté le dénominateur \(\Pr(\text{données})\). Voyons s’il est possible de faire avec. Comme on l’a vu, ce dénominateur est donné par \(\displaystyle \Pr(\text{y}) = \int{\Pr(\text{données} \mid \theta) \Pr(\theta) \, d\theta}\). On va donc devoir calculer cette intégrale. Commençons par écrire une fonction R qui calcule le produit de la vraisemblance (binomiale) par le prior (Beta(1,1)), c’est-à-dire le numérateur dans le théorème de Bayes \(\Pr(\text{données} \mid \theta) \times \Pr(\theta)\) :
Nous pouvons maintenant écrire la fonction qui calcule le dénominateur, et pour ce faire, nous allons utiliser la fonction integrate de R qui permet de calculer l’intégrale d’une fonction d’une variable. La fonction integrate() met en oeuvre des techniques de quadrature pour diviser en petits carrés l’aire sous la courbe délimitée par la fonction à intégrer, et les compter.
den <- integrate(num, 0, 1)$valueNous obtenons alors une approximation numérique de la distribution a posteriori de la survie hivernale comme dans la Figure 2.1 :
# Crée une grille de valeurs possibles pour la probabilité de survie (entre 0 et 1)
grid <- seq(0, 1, 0.01)
# Calcule les valeurs de la densité a posteriori sur la grille
# num(grid) est la vraisemblance * prior, et den est la constante de normalisation
posterior <- data.frame(
survival = grid,
ratio = num(grid) / den # densité a posteriori normalisée
)
# Trace la courbe de la densité a posteriori
posterior %>%
ggplot(aes(x = survival, y = ratio)) +
geom_line(size = 1.5) +
labs(x = "Probabilité de survie", y = "Densité") +
theme_minimal()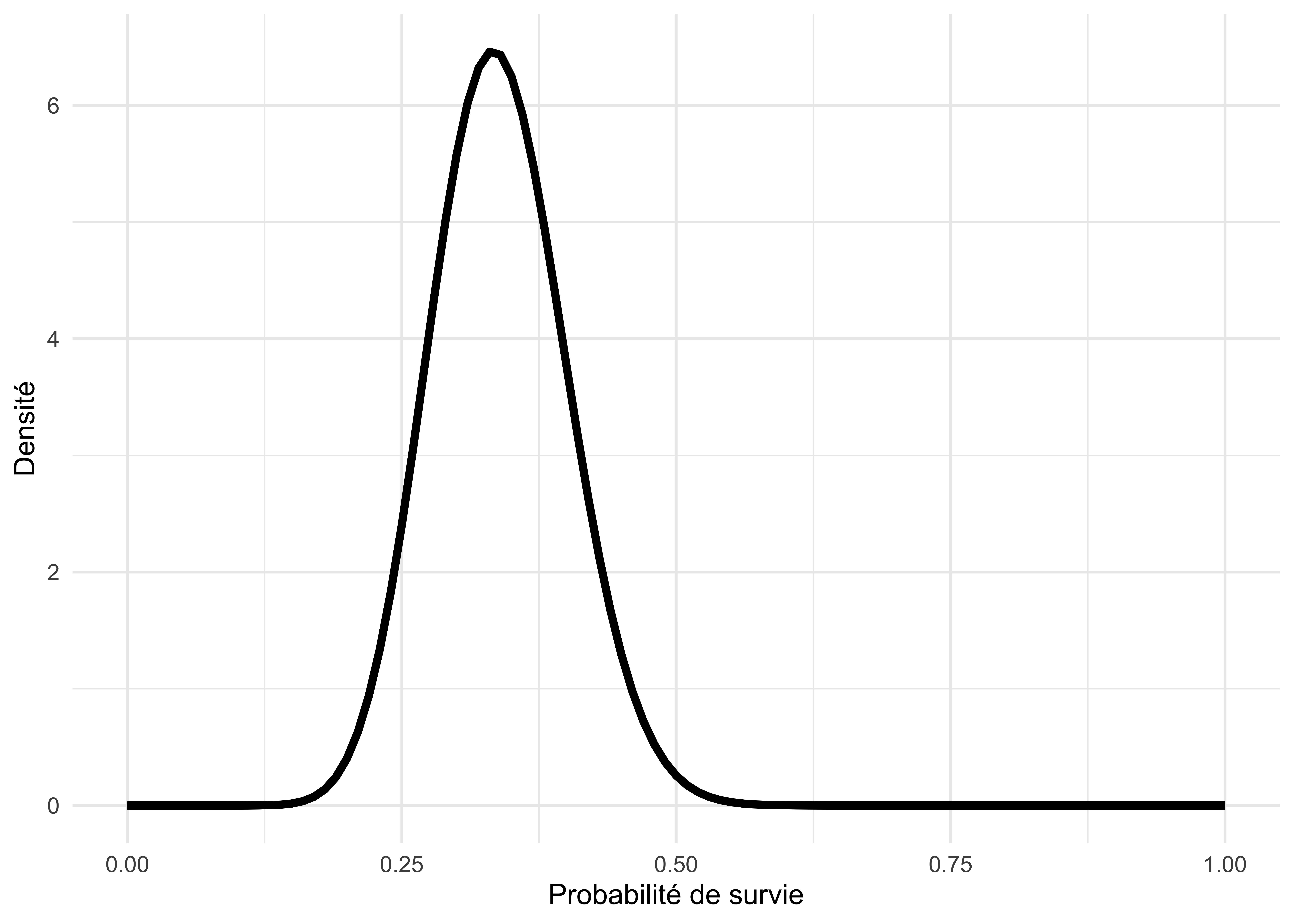
Figure 2.1: Approximation numérique de la distribution a posteriori de la survie hivernale.
Quelle est la qualité de cette approximation numérique ? Idéalement, on voudrait comparer l’approximation à la véritable distribution postérieure. Ça tombe bien, on l’a obtenue dans le Chapitre 1, il s’agit d’une distribution bêta de paramètres 20 et 39. On peut se rendre compte dans la Figure 2.2 que les deux courbes se superposent parfaitement.
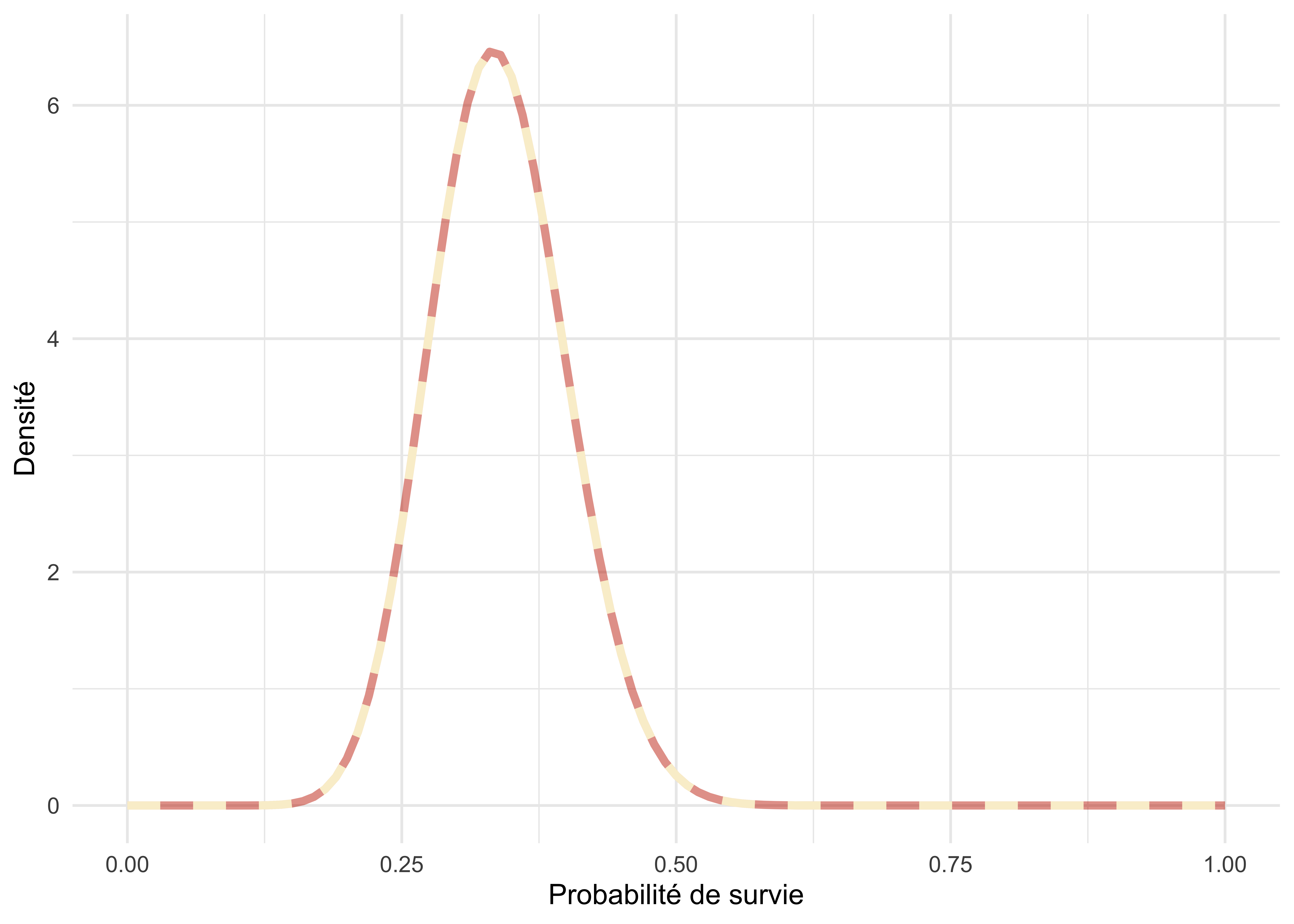
Figure 2.2: Comparaison entre la postérieure exacte (couleur rouge brique) et l’approximation numérique (couleur crème).
La distribution postérieure exacte (couleur rouge brique) et l’approximation numérique (couleur crème) de la survie hivernale ne peuvent être distinguées, ce qui suggère que l’approximation numérique est plus que satisfaisante.
Dans notre exemple, nous avons un seul paramètre à estimer : la survie hivernale. Cela signifie qu’il s’agit d’une intégrale unidimensionnelle au dénominateur, ce qui est assez facile avec les techniques de quadrature et la fonction R integrate().
Mais que se passe-t-il si nous avons plusieurs paramètres ? Par exemple, imaginez que vous vouliez ajuster un modèle de régression avec une probabilité de survie qui dépend d’une variable explicative, par exemple la masse des ragondins. L’effet de cette variable est capturé par le paramètre de régression \(\beta_0\) (l’ordonnée à l’origine), \(\beta_1\) la pente associée, et on a aussi l’erreur résiduelle avec l’écart-type \(\sigma\) (voir Chapitre 5). Le théorème de Bayes donne alors la distribution postérieure jointe de ces paramètres (c’est-à-dire des trois paramètres ensemble) :
\[ \displaystyle \Pr(\beta_0, \beta_1, \sigma \mid \text{y}) = \frac{ \Pr(\text{y} \mid \beta_0, \beta_1, \sigma) \times \Pr(\beta_0, \beta_1, \sigma)}{\displaystyle \iiint \Pr(\text{y} \mid \beta_0, \beta_1, \sigma) \Pr(\beta_0, \beta_1, \sigma) \, d\beta_0 \, d\beta_1 \, d\sigma} \]
Il y a deux défis numériques majeurs :
- Souhaite-t-on vraiment calculer une intégrale triple ? Non, car les méthodes classiques ne vont pas beaucoup plus loin que deux dimensions.
- On s’intéresse souvent aux distributions marginales des paramètres (par exemple celle de \(\beta_1\) correspondant à l’effet de la masse sur la survie), obtenues en intégrant la distribution a posteriori jointe sur les autres paramètres (ici une intégrale double par rapport à \(\beta_0\) et \(\sigma\)) - ce qui devient vite incalculable quand leur nombre augmente.
Dans la section suivante, nous introduisons des méthodes de simulation puissantes pour surmonter ces limitations.
2.3 Les algorithmes MCMC
En bref, l’idée des méthodes de Monte Carlo par chaînes de Markov (MCMC) est d’utiliser des simulations pour approximer les distributions a posteriori avec une certaine précision, en tirant un grand nombre d’échantillons. Cela évite le calcul explicite des intégrales multidimensionnelles auxquelles on a à faire lorsqu’on applique le théorème de Bayes.
Ces algorithmes de simulation se composent de deux parties : chaînes de Markov et Monte Carlo. Essayons de comprendre ces deux termes.
Que signifie Monte Carlo ? L’intégration Monte Carlo est une technique de simulation utilisée pour calculer des intégrales de fonctions quelconques \(f\) d’une variable aléatoire \(X\) suivant une distribution \(\Pr(X)\), comme \(\displaystyle \int f(X) \Pr(X) dX\). On tire des valeurs \(X_1, \ldots, X_k\) dans \(\Pr(X)\), on applique la fonction \(f\) à ces valeurs, puis on calcule la moyenne de ces nouvelles valeurs : \(\displaystyle{\frac{1}{k}}\sum_{i=1}^k{f(X_i)}\) pour approximer l’intégrale.
Comment utilise-t-on l’intégration Monte Carlo dans un contexte bayésien ? La distribution a posteriori contient toute l’information nécessaire sur le paramètre à estimer. Mais lorsqu’il y a plusieurs paramètres, on souhaite souvent résumer cette information en calculant des résumés numériques. Le résumé le plus simple est la moyenne de la distribution a posteriori, soit \(E(\theta) = \int \theta \Pr(\theta \mid \text{données}) \, d\theta\), où \(X\) est ici \(\theta\) et \(f\) est l’identité. Cette moyenne a posteriori peut être estimée par intégration Monte Carlo ; par exemple, pour la survie des ragondins :
# tirage de 1000 valeurs depuis la postérieure bêta(20,39)
sample_from_posterior <- rbeta(1000, 20, 39)
# calcul de la moyenne par intégration Monte Carlo
mean(sample_from_posterior)
#> [1] 0.3390116On peut vérifier que la moyenne obtenue est proche de l’espérance théorique d’une distribution bêta :
20/(20+39) # espérance de la loi bêta(20,39)
#> [1] 0.3389831Un autre résumé numérique utile est l’intervalle de crédibilité à l’intérieur duquel se trouve le paramètre avec une certaine probabilité, généralement 0.95, soit un intervalle de crédibilité à 95%. Déterminer les bornes d’un tel intervalle nécessite le calcul de quantiles, ce qui implique aussi des intégrales, donc un recours à l’intégration Monte Carlo. Un intervalle de crédibilité à 95% pour la survie hivernale peut être obtenu avec :
En passant, il y a une différence entre l’intervalle de crédibilité en statistique bayésienne et l’intervalle de confiance de la statistique fréquentiste. Un intervalle de confiance à 95% signifie que si l’on répétait l’expérience un très grand nombre de fois (équiper des ragondins de GPS et constater le nombre de survivants à l’hiver), environ 95% des intervalles construits de cette manière contiendraient la vraie valeur \(\theta\) du paramètre. Mais on ne peut pas dire que la probabilité que le paramètre soit dans un intervalle donné est de 95%. Un intervalle de crédibilité à 95%, en revanche, signifie qu’il y a 95% de probabilité que le paramètre se trouve dans cet intervalle. L’interprétation de l’intervalle de crédibilité est un peu plus intuitive que celle de l’intervalle de confiance.
Maintenant, qu’est-ce qu’une chaîne de Markov ? Une chaîne de Markov est une séquence aléatoire de nombres, dans laquelle chaque nombre dépend uniquement du nombre précédent. Un exemple est la météo dans ma ville, Montpellier, dans le sud de la France, où une journée ensoleillée est très probablement suivie d’une autre journée ensoleillée, disons avec une probabilité de 0.8, et une journée pluvieuse est rarement suivie d’une autre journée pluvieuse, disons avec une probabilité de 0.1. La dynamique de cette chaîne de Markov est capturée par la matrice de transition :
\[ \begin{array}{c|cc} & \text{Ensoleillé demain} & \text{Pluvieux demain} \\\\ \hline \text{Ensoleillé aujourd'hui} & 0.8 & 0.2 \\\\ \text{Pluvieux aujourd'hui} & 0.9 & 0.1 \end{array} \]
Les lignes indiquent la météo aujourd’hui, et les colonnes, celle de demain. Les cellules donnent la probabilité d’avoir une journée ensoleillée ou pluvieuse demain, selon le temps qu’il fait aujourd’hui (des probabilités conditionnelles, voir Chapitre 1).
Sous certaines conditions, une chaîne de Markov converge vers une distribution stationnaire unique. Dans notre exemple météorologique, lançons la chaîne pour 20 étapes :
temps <- matrix(c(0.8, 0.2, 0.9, 0.1), nrow = 2, byrow = T) # matrice de transition
etapes <- 20
for (i in 1:etapes){
temps <- temps %*% temps # multiplication matricielle
}
round(temps, 2) # produit matriciel après 20 étapes
#> [,1] [,2]
#> [1,] 0.82 0.18
#> [2,] 0.82 0.18Chaque ligne de la matrice converge vers la même distribution \((0.82, 0.18)\) au fur et à mesure que le nombre d’étapes augmente. La convergence se produit quel que soit l’état de départ : on a alors une probabilité de 0.82 d’avoir du soleil et de 0.18 d’avoir de la pluie.
Revenons aux méthodes MCMC. L’idée centrale est que l’on peut construire une chaîne de Markov dont la distribution stationnaire est justement la distribution a posteriori de nos paramètres. Retenez bien cette idée, elle est centrale.
En combinant Monte Carlo et chaînes de Markov, les méthodes MCMC nous permettent de générer un échantillon de valeurs dont la distribution converge vers la distribution a posteriori (chaîne de Markov), et d’utiliser cet échantillon pour calculer des résumés numériques a posteriori (Monte Carlo), comme la moyenne ou les intervalles de crédibilité.
Il existe plusieurs manières de construire des chaînes de Markov pour l’inférence bayésienne. Vous avez peut-être entendu parler de l’algorithme de Metropolis-Hastings ou de l’échantillonneur de Gibbs. Vous pouvez consulter https://chi-feng.github.io/mcmc-demo/ pour une galerie interactive d’algorithmes MCMC. Ici, j’illustre l’algorithme de Metropolis et sa mise en œuvre pratique. Pour cela je m’inspire de l’excellent livre de Jim Albert (2009). Il n’est pas question d’être capable d’écrire un tel algorithme par soi-même, seulement d’en saisir les grandes lignes et, surtout, la notion de simulations.
Revenons à notre exemple d’estimation de la survie. Nous allons illustrer l’échantillonnage depuis la distribution a posteriori de la survie. Commençons par écrire les fonctions pour la vraisemblance, le prior et la postérieure. On se place sur l’échelle log pour manipuler des sommes et des soustractions plutôt que des produits et des ratios qui rendent les calculs numériques instables :
# 19 animaux retrouvés vivants sur 57 capturés, marqués et relâchés
y <- 19
n <- 57
# log-vraisemblance binomiale Bin(n = 57,p)
loglikelihood <- function(x, p){
dbinom(x = x, size = n, prob = p, log = TRUE)
}
# densité du prior uniforme
logprior <- function(p){
dunif(x = p, min = 0, max = 1, log = TRUE)
# ou bien dbeta(x = p, shape1 = 0, shape2 = 1, log = TRUE)
}
# densité a posteriori (échelle logarithmique)
posterior <- function(x, p){
loglikelihood(x, p) + logprior(p)
}L’algorithme de Metropolis fonctionne comme suit :
On choisit une valeur initiale pour le paramètre à estimer. C’est notre valeur de départ, ou point initial de la chaîne de Markov.
Pour décider de l’étape suivante, on propose de s’éloigner de la valeur courante du paramètre — c’est la valeur candidate. On ajoute à la valeur courante une valeur tirée d’une loi normale avec une certaine variance — c’est la loi de proposition. L’algorithme de Metropolis est un cas particulier de celui de Metropolis-Hastings avec des propositions symétriques.
On calcule le rapport des vraisemblances entre la position candidate et la position courante : \(R = \displaystyle \frac{\Pr(\text{valeur candidate}|\text{données})}{\Pr(\text{valeur courante}|\text{données})}\). Pour calculer le numérateur et le dénominateur, il suffit d’appliquer le théorème de Bayes, et c’est là que la magie des méthodes MCMC opère car, comme la quantité \(\Pr(\text{données})\) apparaît au numérateur et au dénominateur, elle s’annule et plus besoin de la calculer ! On a remplacé le calcul d’une intégrale par des simulations.
Si la densité postérieure en la position candidate est plus grande qu’en la position courante, autrement dit si la valeur candidate est plus plausible, on l’accepte sans hésiter. Sinon, on l’accepte avec probabilité \(R\), et on la rejette avec probabilité \(1 - R\). Par exemple, si la valeur candidate est dix fois moins plausible, on l’accepte avec probabilité 0.1. On utilise un générateur uniforme entre 0 et 1 (appelons-le \(X\)), et si \(X < R\), on accepte la valeur candidate, sinon on reste à la valeur courante. En pratique, on vise un taux d’acceptation entre 0.2 et 0.4 qu’on peut ajuster en calibrant la variance de la proposition, cela permet d’explorer tout le champ des possibles.
On répète les étapes 2 à 4 un certain nombre de fois — ce sont les itérations.
Assez de théorie, passons à l’implémentation. On commence par initialiser :
steps <- 100 # nombre d'étapes ou itérations de la chaîne
theta.post <- rep(NA, steps) # vecteur pour stocker les valeurs simulées
accept <- rep(NA, steps) # vecteur pour enregistrer les acceptations/rejets
set.seed(666) # pour la reproductibilitéPourquoi faut-il initialiser ? Avant de lancer la chaîne de Markov, on prépare les objets dans lesquels seront stockées les valeurs simulées de notre paramètre (ici, la probabilité de survie), ainsi que l’information sur l’acceptation ou non de chaque proposition. Et set.seed(666), à quoi ça sert ? Cette commande fixe la graine du générateur de nombres aléatoires. Elle permet de garantir que les simulations soient reproductibles : en relançant le code, vous obtiendrez exactement les mêmes valeurs simulées que les miennes.
On choisit une valeur de départ :
inits <- 0.5 # valeur de départ choisie pour theta
theta.post[1] <- inits # on enregistre cette valeur comme première position de la chaîne
accept[1] <- 1 # la valeur initiale est acceptée par défautPourquoi une valeur de départ ? Une chaîne de Markov doit bien commencer quelque part : ici, on choisit arbitrairement 0.5 comme valeur initiale de la probabilité de survie. La seule contrainte est que cette valeur soit compatible avec le prior, on ne va pas prendre une survie négative ou de 15. On place cette valeur dans le premier élément du vecteur theta.post, et on indique dans accept[1] <- 1 que cette première valeur est acceptée par construction, puisque c’est notre point de départ.
Puis, on écrit une fonction pour proposer une valeur candidate à partir de la valeur courante :
move <- function(x, away = 1){
logitx <- log(x / (1 - x)) # transformation logit : transforme x de (0,1) vers (-∞,+∞)
logit_candidate <- logitx + rnorm(1, 0, away) # on ajoute un bruit normal centré, de variance contrôlée par away
candidate <- plogis(logit_candidate) # transformation réciproque (logit^-1) : retourne une valeur entre 0 et 1
return(candidate) # retourne la valeur proposée
}Cette fonction introduit une proposition aléatoire autour de la valeur courante. On travaille sur l’échelle logit pour s’assurer que la proposition finale (candidate) reste toujours dans l’intervalle (0,1) (voir aussi le Chapitre 6). Le paramètre away contrôle la dispersion des propositions : plus il est grand, plus la chaîne pourra faire de grands sauts ; plus il est petit, plus les propositions seront proches de la valeur actuelle.
Ensuite, on applique les étapes 2 à 4 de l’algorithme dans une boucle (c’est l’étape 5, répétition des itérations) :
for (t in 2:steps){ # pour chaque itération, à partir de la 2e
# Étape 2 : proposer une nouvelle valeur pour theta
theta_star <- move(theta.post[t-1]) # valeur candidate tirée à partir de la valeur précédente
# Étape 3 : calculer le rapport des densités postérieures (échelle log)
pstar <- posterior(y, p = theta_star) # densité a posteriori à la valeur candidate
pprev <- posterior(y, p = theta.post[t-1]) # densité a posteriori à la valeur courante
logR <- pstar - pprev # différence sur l’échelle log
R <- exp(logR) # on revient à l’échelle naturelle (rapport des densités)
# Étape 4 : décider si on accepte ou rejette la proposition
X <- runif(1, 0, 1) # tirage aléatoire entre 0 et 1 : la "roulette" d’acceptation
if (X < R){ # si la proposition est plus plausible (ou pas trop pire)
theta.post[t] <- theta_star # on accepte et on stocke la valeur candidate
accept[t] <- 1 # on note que la proposition a été acceptée
} else {
theta.post[t] <- theta.post[t-1] # sinon on reste sur la valeur précédente
accept[t] <- 0 # on note le refus
}
}Cette boucle construit itérativement la chaîne de Markov. La probabilité d’accepter une valeur moins plausible est proportionnelle à son rapport de vraisemblance. Le vecteur accept permet ensuite de diagnostiquer la fréquence d’acceptation, utile pour calibrer la chaîne.
Jetons un coup d’oeil aux premières et dernières valeurs simulées :
head(theta.post)
#> [1] 0.5000000 0.5000000 0.3021903 0.3021903 0.1853669 0.1853669
tail(theta.post)
#> [1] 0.4076667 0.4076667 0.4076667 0.4076667 0.2914464 0.2914464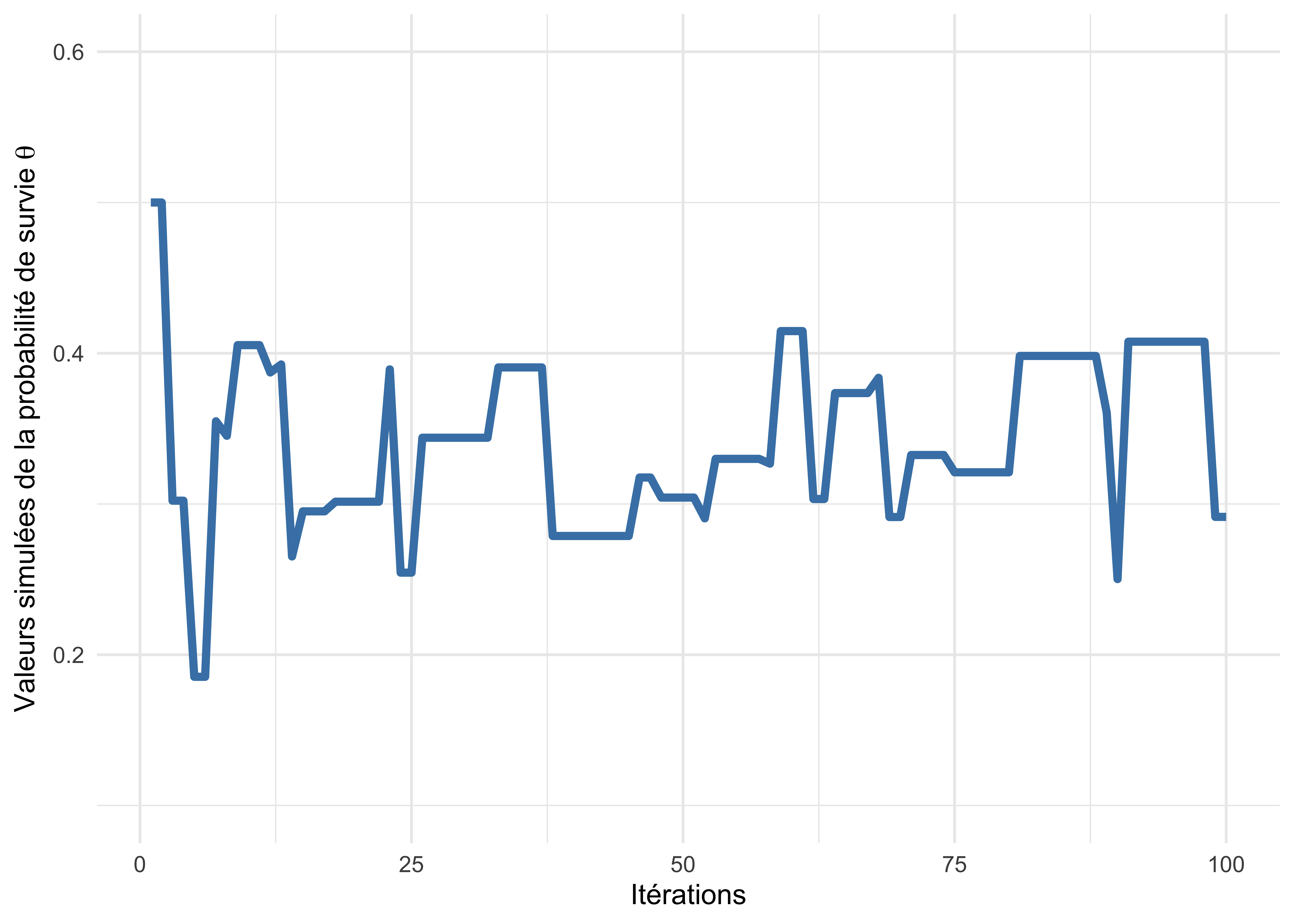
Figure 2.3: Trace plot des valeurs simulées de la probabilité de survie \(\theta\) au fil des itérations.
Que nous apprend ce trace plot ? L’axe horizontal représente les itérations (ou temps dans la chaîne de Markov). L’axe vertical montre les valeurs simulées de la probabilité de survie à chaque étape. Dans la figure, on observe que la chaîne reste parfois pendant plusieurs itérations consécutives à la même valeur. Cela se produit lorsque la valeur candidate proposée par l’algorithme est rejetée – la chaîne conserve alors la valeur précédente (ou courante plus précisément). À d’autres moments, on voit des sauts vers de nouvelles valeurs, qui correspondent aux propositions acceptées.
On peut ensuite encapsuler l’algorithme dans une fonction réutilisable, ce qui permet de lancer facilement plusieurs chaînes :
metropolis <- function(steps = 100, inits = 0.5, away = 1){
theta.post <- rep(NA, steps) # on crée un vecteur pour stocker les échantillons
theta.post[1] <- inits # on initialise avec la valeur de départ
for (t in 2:steps){ # boucle sur les étapes (à partir de la 2ème)
theta_star <- move(theta.post[t-1], away) # proposition d'une nouvelle valeur
# on calcule le log-ratio de la densité a posteriori entre candidat et position courante
logR <- posterior(y, theta_star) -
posterior(y, theta.post[t-1])
R <- exp(logR) # passage à l'échelle normale (non log)
X <- runif(1, 0, 1) # tirage d'un nombre aléatoire uniforme
theta.post[t] <- ifelse(X < R, # si le tirage < probabilité d'acceptation...
theta_star, # ... on accepte la valeur proposée
theta.post[t-1]) # sinon on conserve la précédente
}
return(theta.post) # on retourne l’échantillon simulé
}On peut maintenant utiliser la fonction metropolis() pour lancer une autre chaîne, cette fois-ci en partant de 0.2 :
theta.post2 <- metropolis(steps = 100, inits = 0.2) # départ à 0.2Notez qu’on parle souvent de “lancer plusieurs chaînes” MCMC afin de diagnostiquer la convergence. Il s’agit en réalité de réalisations indépendantes de la même chaîne de Markov, comme si on lançait plusieurs fois une pièce avec une distribution un peu plus compliquée qu’une Bernoulli.
On trace ensuite les deux chaînes ensemble comme dans la Figure 2.4 :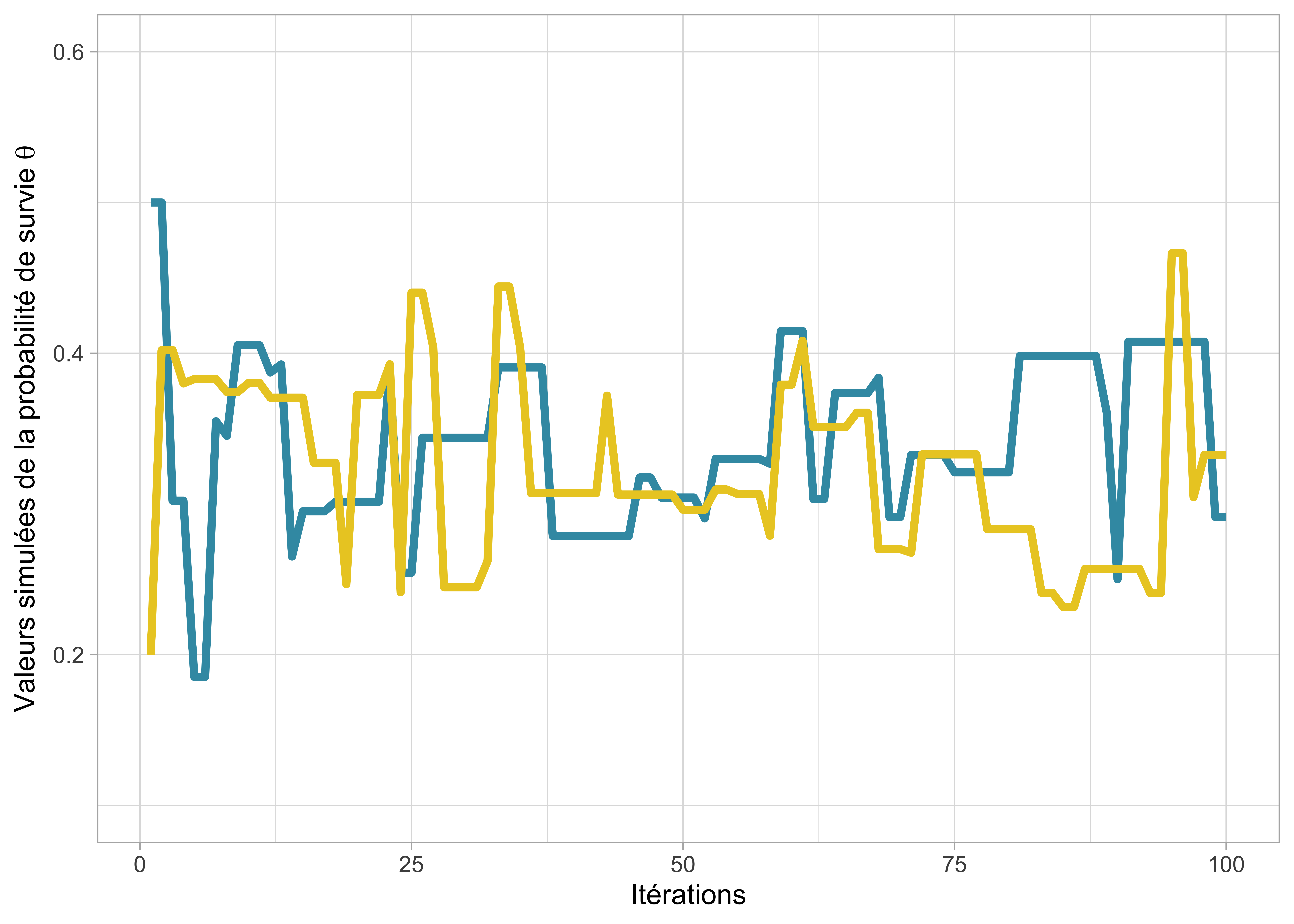
Figure 2.4: Trace plot des valeurs simulées de la probabilité de survie \(\theta\) au fil des itérations. Deux chaînes ont été lancées avec des valeurs initiales différentes, 0.5 en bleu et 0.2 en jaune.
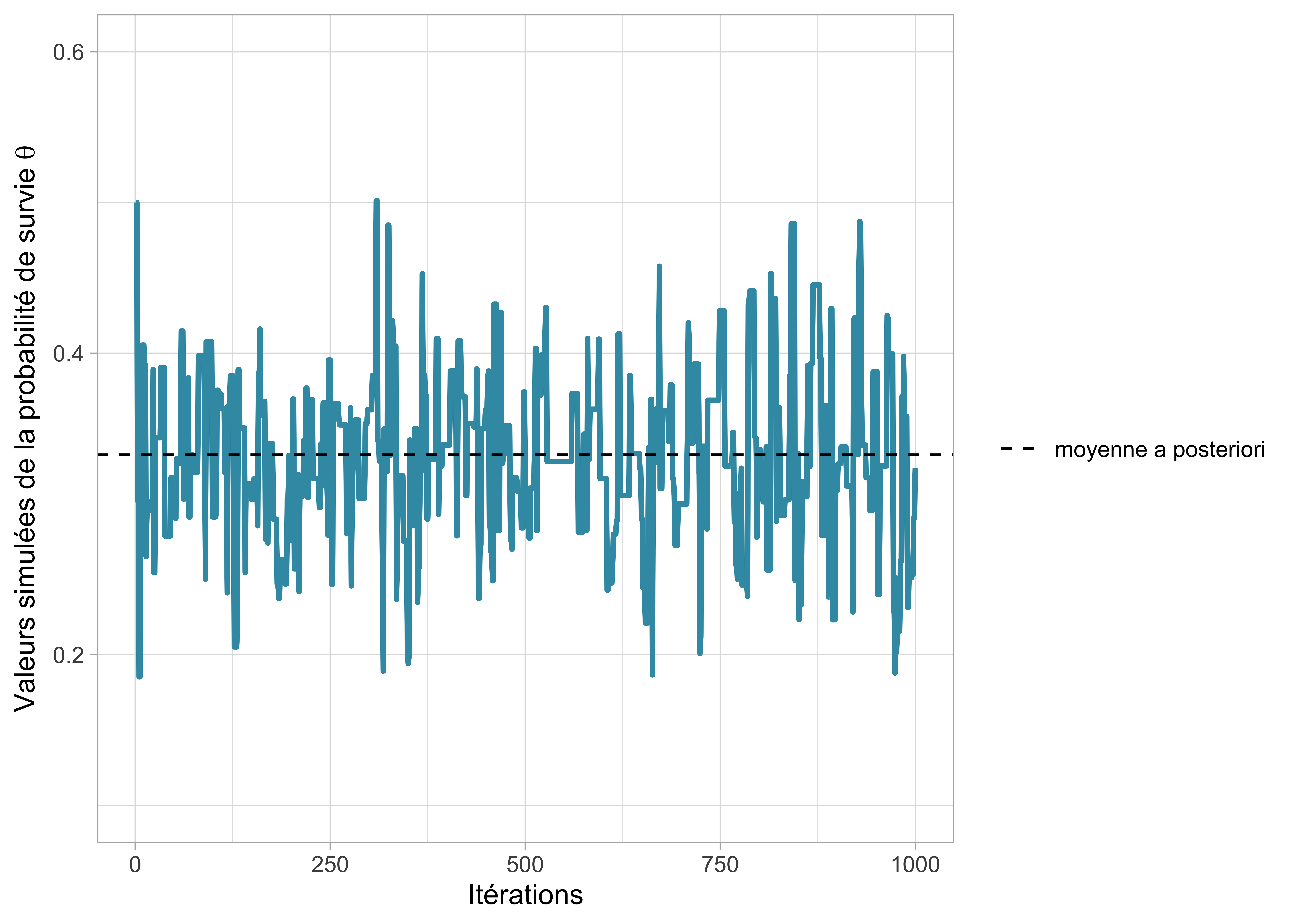
Figure 2.5: Trace plot des valeurs simulées de la probabilité de survie \(\theta\) au fil des 1000 itérations.
Avec un grand nombre d’itérations, chaque chaîne devrait se stabiliser autour de sa distribution stationnaire. Visuellement, on recherche une zone dense, homogène et bien explorée, ressemblant à une pelouse bien tondue (c’est une image).
Une fois la distribution stationnaire atteinte, vous pouvez considérer les valeurs simulées de la chaîne de Markov comme un échantillon de la distribution a posteriori et obtenir des résumés numériques des paramètres (moyenne a posteriori, intervalle de crédibilité).
Quand peut-on dire qu’on a atteint cette distribution stationnaire ? Une fois qu’on a la convergence, combien de simulations faut-il encore faire pour obtenir une bonne approximation de la distribution a posteriori de nos paramètres ? Je réponds à ces questions dans la section suivante.
2.4 Évaluer la convergence
Lorsqu’on applique une méthode MCMC, il faut déterminer combien de temps il faut à la chaîne de Markov pour converger vers la distribution cible, et combien d’itérations supplémentaires sont nécessaires après la convergence pour obtenir des estimations Monte Carlo fiables des résumés numériques (moyennes a posteriori, intervalles de crédibilité).
2.4.1 Burn-in
En pratique, on ignore les premières valeurs de la chaîne de Markov et on n’utilise que les valeurs simulées après convergence. Les observations initiales que l’on écarte sont généralement appelées la période de burn-in (ou pré-chauffage).
La méthode la plus simple pour déterminer la durée de la période de burn-in est d’inspecter les trace plots. Reprenons notre exemple et observons dans la Figure 2.6 un trace plot pour une chaîne démarrant à la valeur 0.99 :
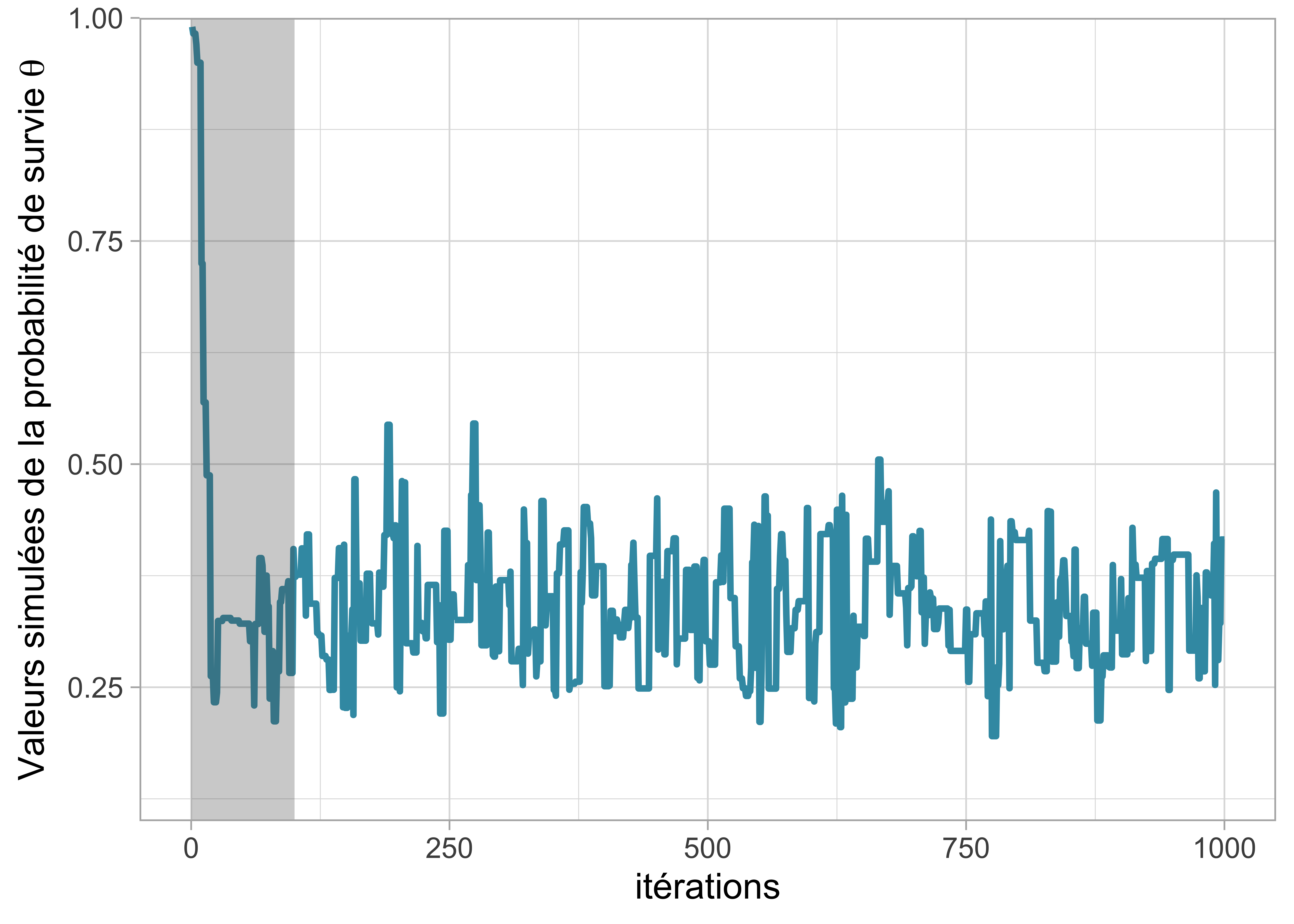
Figure 2.6: Trace plot pour une chaîne démarrant à 0.99. La zone ombrée illustre une période de burn-in possible.
La chaîne démarre à 0.99 et se stabilise rapidement, les valeurs oscillant autour de 0.3 à partir de la 100ème itération. On peut choisir la zone ombrée comme période de burn-in et éliminer les 100 premières valeurs. Par prudence, on pourrait utiliser 250 voire 500 itérations comme burn-in, si ça ne coûte pas trop cher en temps de calcul évidemment.
Examiner un trace plot d’une seule chaîne est utile, mais on lance généralement plusieurs chaînes avec des valeurs initiales différentes pour vérifier que toutes atteignent la même distribution stationnaire. Cette approche est formalisée par la statistique de Brooks-Gelman-Rubin (BGR), notée \(\hat{R}\), qui mesure le ratio entre la variabilité totale (entre les chaînes, plus à l’intérieur de chaque chaîne) et la variabilité intra-chaîne. Elle est proche du test \(F\) dans une analyse de variance (ici à un facteur dont les modalités seraient les chaînes). Une valeur inférieure à 1.1 indique une convergence probable.
Revenons à notre exemple : nous exécutons deux chaînes de Markov avec des valeurs initiales de 0.2 et 0.8, en faisant varier le nombre d’itérations de 100 à 1000 toutes les 50 itérations, et nous calculons la statistique BGR en utilisant la moitié des itérations comme période de burn-in (Figure 2.7).
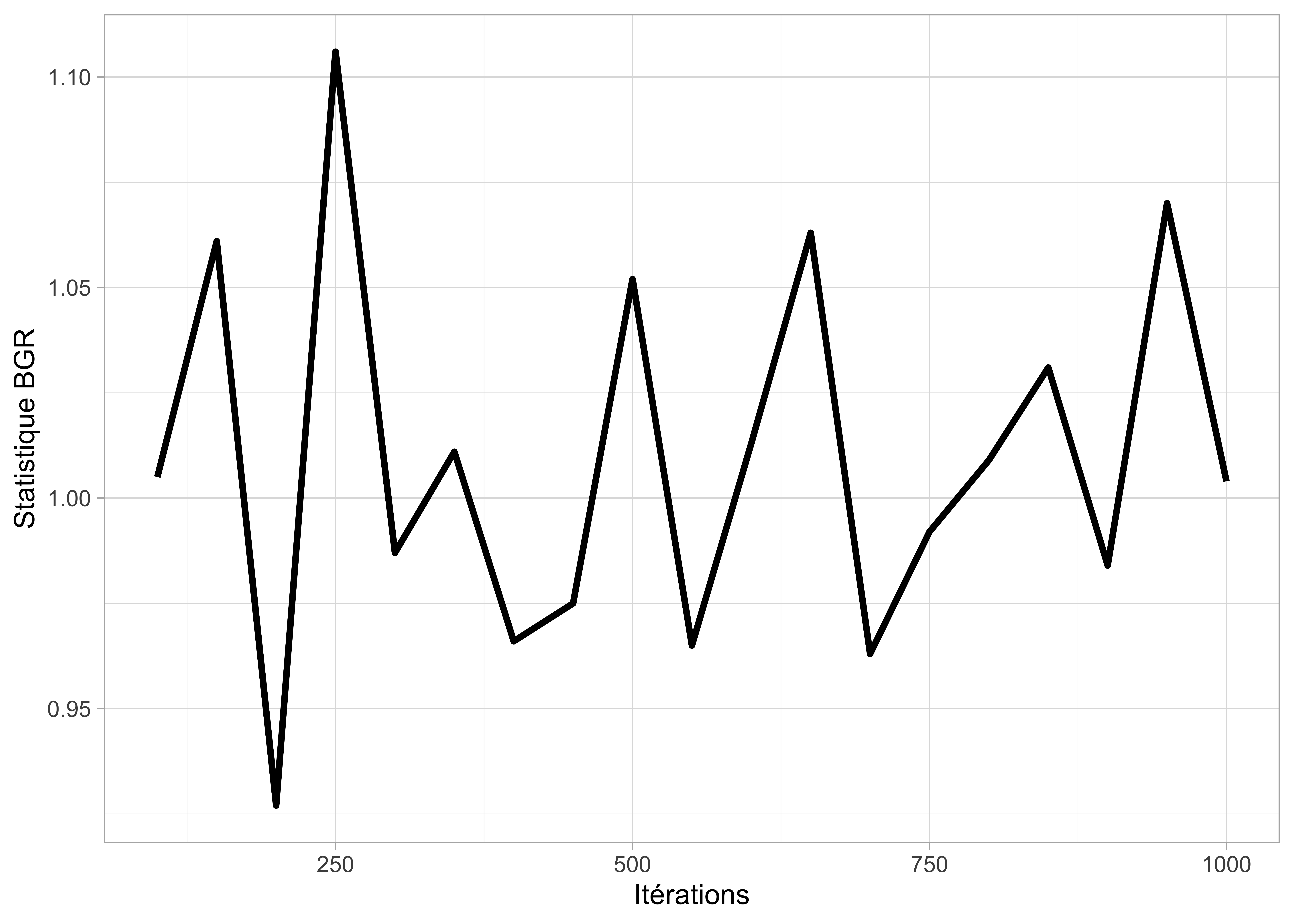
Figure 2.7: Valeur de la statistique de Brooks-Gelman-Rubin (BGR) en fonction du nombre d’itérations. Une valeur proche de 1 suggère la convergence.
Nous obtenons une valeur de la statistique BGR proche de 1 dès 300 itérations, ce qui suggère qu’avec un burn-in de 300 itérations, rien n’indique un problème de convergence.
Il est important de garder à l’esprit qu’une valeur proche de 1 pour la statistique BGR constitue une condition nécessaire, mais non suffisante, à la convergence. En d’autres termes, ce diagnostic ne permet pas d’affirmer avec certitude que la chaîne a convergé, mais simplement qu’on ne détecte pas de signe évident qu’elle ne l’a pas fait. Mon conseil : prenez toujours le temps de jeter un coup d’oeil aux trace plots.
2.4.2 Longueur de chaîne
Quelle longueur de chaîne est nécessaire pour obtenir des estimations fiables des paramètres ? Il faut garder à l’esprit que les étapes successives d’une chaîne de Markov ne sont pas indépendantes. C’est ce qu’on appelle l’autocorrélation. Idéalement, on cherche à minimiser cette autocorrélation.
Ici encore, les trace plots permettent de diagnostiquer des problèmes d’autocorrélation. Revenons à l’exemple de survie. La Figure 2.8 ci-dessous montre les trace plots (3000 itérations) pour différentes valeurs de l’écart-type (paramètre away) de la loi normale de proposition utilisée pour générer les valeurs candidates.
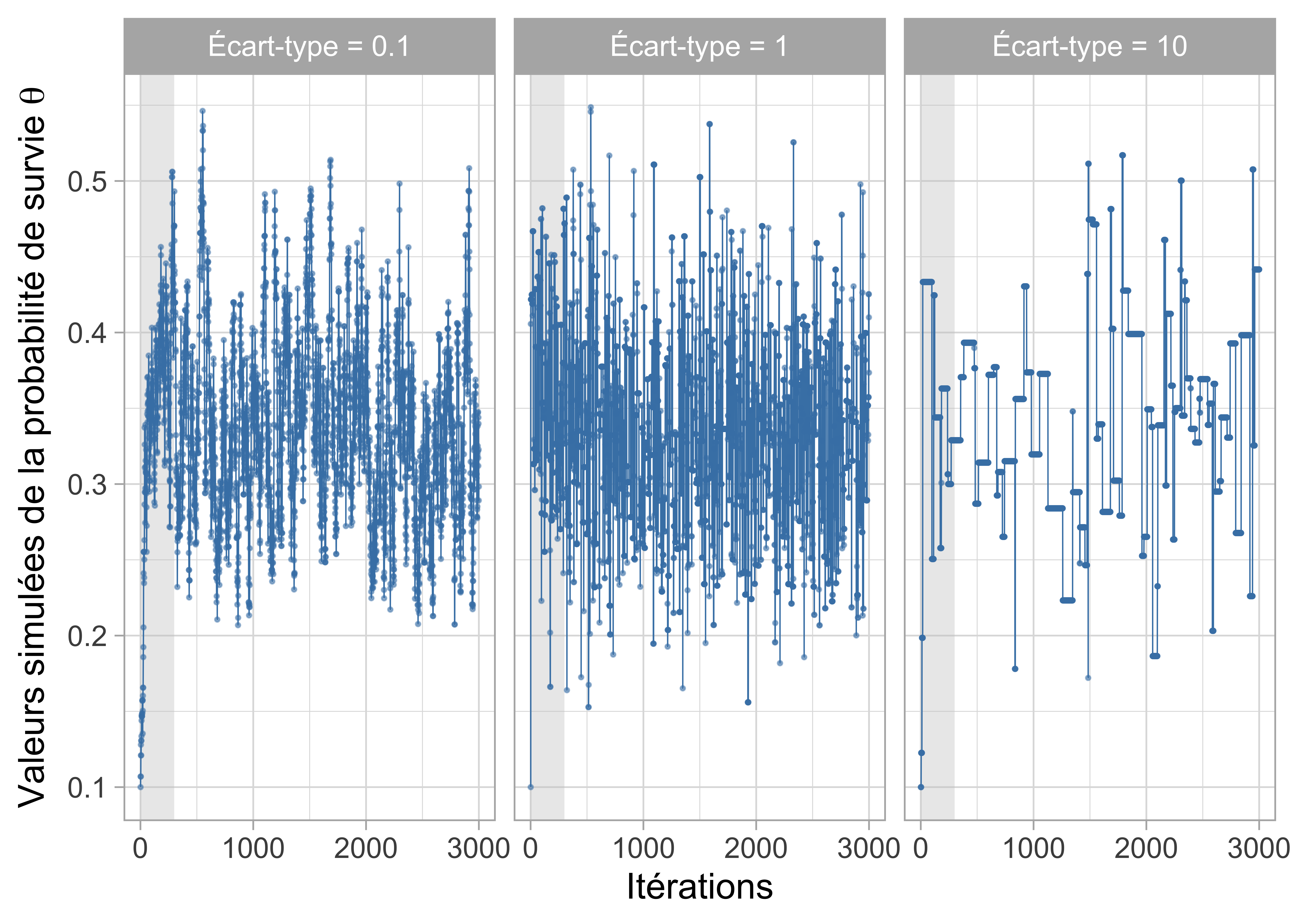
Figure 2.8: Trace plots pour différentes valeurs de l’écart-type de la proposition (away). Un bon mixing est observé avec away = 1. La zone grise ombrée correspond à un burn-in de 300 itérations.
Les petits et grands déplacements visibles dans les panneaux de gauche et de droite entraînent une forte corrélation entre les observations successives de la chaîne de Markov, tandis qu’un écart-type égal à 1 (au centre) permet une exploration efficace de l’espace des paramètres. Ce mouvement dans l’espace des paramètres est appelé “mixing”. Le mixing est considéré comme mauvais lorsque la chaîne fait de trop petits ou de trop grands sauts, et comme bon dans le cas contraire.
En complément des trace plots, les graphiques de la fonction d’autocorrélation (ACF) offrent un moyen pratique de visualiser la force de l’autocorrélation dans un échantillon donné. Les ACF montrent la corrélation entre les valeurs échantillonnées successivement, séparées par un nombre croissant d’itérations, appelé lag (décalage). On obtient dans la Figure 2.9 ces graphiques de fonction d’autocorrélation pour différentes valeurs de l’écart-type de la distribution de proposition grâce à la fonctionforecast::ggAcf() :
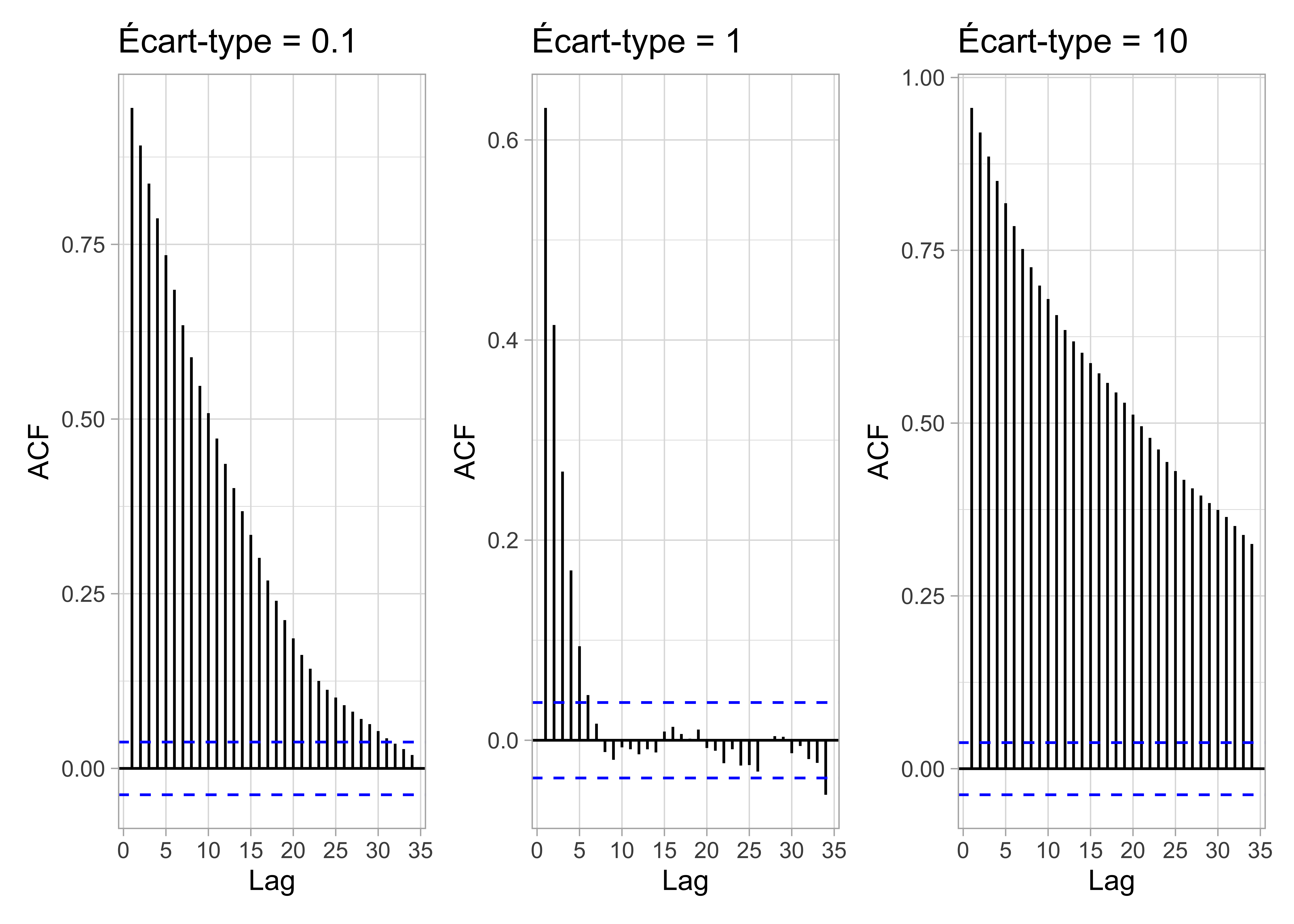
Figure 2.9: Fonctions d’autocorrélation (ACF) pour différentes valeurs de l’écart-type de la proposition. Une faible autocorrélation est un signe de bon mixing. Un burn-in de 300 itérations est appliqué.
Dans les panneaux de gauche et de droite, l’autocorrélation est forte, diminue lentement avec le lag, et le mixing est mauvais. Dans le panneau central, l’autocorrélation est faible, diminue rapidement avec le lag, et le mixing est bon.
L’autocorrélation n’est pas forcément un gros problème. Des observations fortement corrélées nécessitent simplement un plus grand nombre d’échantillons, et donc des simulations plus longues. Mais combien d’itérations faut-il exactement ? La taille effective de l’échantillon (n.eff) mesure la longueur utile de la chaîne en tenant compte de l’autocorrélation. Il est recommandé de vérifier la n.eff pour chaque paramètre d’intérêt, ainsi que pour toute combinaison pertinente de paramètres. En général, on considère qu’il faut au moins \(\text{n.eff} \geq 400\) observations indépendantes pour obtenir des estimations Monte Carlo fiables des paramètres du modèle. Dans l’exemple de la survie animale, n.eff peut être calculée avec la fonction effectiveSize() du package coda:
# Générer les chaînes pour trois valeurs d'écart-type
d <- tibble(away = c(0.1, 1, 10)) %>%
mutate(accepted_traj = map(away,
metropolis,
steps = n_steps,
inits = 0.1)) %>%
unnest(accepted_traj) %>%
mutate(proposal_sd = str_c("Écart-type = ", away),
iter = rep(1:n_steps, times = 3))
# Calculer la taille effective d'échantillon
neff1 <- coda::effectiveSize(d$accepted_traj[d$proposal_sd=="Écart-type = 0.1"][-c(1:300)])
neff2 <- coda::effectiveSize(d$accepted_traj[d$proposal_sd=="Écart-type = 1"][-c(1:300)])
neff3 <- coda::effectiveSize(d$accepted_traj[d$proposal_sd=="Écart-type = 10"][-c(1:300)])
tibble("Écart-type" = c(0.1, 1, 10),
"n.eff" = round(c(neff1, neff2, neff3)))
#> # A tibble: 3 × 2
#> `Écart-type` n.eff
#> <dbl> <dbl>
#> 1 0.1 81
#> 2 1 524
#> 3 10 77Comme on pouvait s’y attendre, la valeur de n.eff est inférieure au nombre total d’itérations MCMC (3000) en raison de l’autocorrélation. Ce n’est que lorsque l’écart-type de la distribution de proposition est égal à 1 que le mixing est bon (n.eff \(\geq 400\)), ce qui permet d’obtenir une taille d’échantillon effective satisfaisante.
2.4.3 Et si vous avez des problèmes de convergence ?
Lorsque vous diagnostiquez la convergence d’une chaîne MCMC, vous rencontrerez (très) souvent des difficultés. Cette section propose quelques conseils pratiques qui, je l’espère, vous seront utiles.
Lorsque le mixing est mauvais et que la taille effective de l’échantillon est faible, il peut suffire d’augmenter la période de burn-in et/ou d’augmenter le nombre de simulations. L’utilisation de priors plus informatifs peut également faciliter la convergence des chaînes de Markov, en aidant l’algorithme MCMC à explorer plus efficacement l’espace des paramètres (Chapitre 4). Dans le même esprit, choisir de meilleures valeurs initiales pour démarrer la chaîne peut aussi améliorer les choses. Une stratégie utile consiste à utiliser les estimations d’un modèle plus simple pour lequel vos chaînes MCMC convergent déjà.
Si les problèmes de convergence persistent, il y a souvent un problème avec le modèle lui-même. Un bug dans le code ? Une faute de frappe ? Une erreur dans les équations ? Comme souvent en programmation, le meilleur moyen d’identifier le problème est de réduire la complexité du modèle et de repartir d’un modèle plus simple, jusqu’à ce que vous trouviez ce qui ne va pas.
Un autre conseil est de considérer votre modèle avant tout comme un générateur de données. Simulez des données à partir de ce modèle, en utilisant des valeurs réalistes pour les paramètres, puis tentez de retrouver ces paramètres en ajustant le modèle aux données simulées. Cette approche vous aidera à mieux comprendre comment le modèle fonctionne, ce qu’il ne fait pas, et la quantité de données nécessaire pour obtenir des estimations de paramètres fiables. On reviendra sur cette technique dans les Chapitres 5 et 6.
2.5 En résumé
L’idée des méthodes de Monte Carlo par chaînes de Markov (MCMC) est de simuler des valeurs à partir d’une chaîne de Markov dont la distribution stationnaire est précisément la distribution a posteriori des paramètres qu’on cherche à estimer.
En pratique, on lance plusieurs chaînes de Markov en partant de valeurs initiales dispersées.
On écarte les premières itérations (phase de pré-chauffage ou burn-in) et on considère que la convergence est atteinte lorsque toutes les chaînes convergent vers le même régime.
À partir de là, on fait tourner les chaînes suffisamment longtemps, puis on calcule des estimations Monte Carlo de résumés numériques (par exemple, les moyennes a posteriori ou les intervalles de crédibilité) des paramètres.
Évidemment, on n’a pas envie de construire et implémenter à la main les méthodes MCMC à chaque nouvelle analyse, et on verra dans le Chapitre 3 comment se faciliter la tâche.