Étudier la démographie et la distribution des espèces en conditions naturelles : les modèles de Markov cachés
O. Gimenez, J. Louvrier, V. Lauret, N. Santostasi
Cas d’étude capture-recapture : estimation de la prévalence
Ajustement du modèle
On ajuste le modèle \((\pi,\phi_{\mbox{état}},p,\delta)\).
On commence par lire les données.
wolf_data <- read.table("dat/wolf_capturerecapture.txt")
wolf_data## V1 V2 V3 V4 V5
## 1 0 1 0 0 0
## 2 1 0 0 0 0
## 3 0 0 0 1 0
## 4 1 3 0 0 3
## 5 1 3 3 0 0
## 6 0 0 1 0 0
## 7 0 0 1 0 0
## 8 0 0 0 1 3
## 9 0 0 0 1 0
## 10 0 1 0 0 0
## 11 0 0 3 3 0
## 12 0 3 0 0 0
## 13 0 0 3 0 0
## 14 3 0 0 0 0
## 15 0 0 0 0 3
## 16 3 3 3 0 0
## 17 0 0 2 0 0
## 18 0 0 0 2 0
## 19 0 0 0 2 3
## 20 0 0 0 2 0
## 21 0 0 0 2 3
## 22 0 0 0 0 2
## 23 2 3 0 3 3
## 24 2 0 0 0 0
## 25 2 3 0 3 0
## 26 0 0 2 0 0
## 27 0 2 3 3 0
## 28 0 0 0 2 3
## 29 2 3 0 3 3
## 30 0 2 3 3 0
## 31 0 2 3 0 0
## 32 0 0 2 0 0
## 33 0 0 2 0 0
## 34 0 0 2 0 0
## 35 0 0 2 0 0
## 36 2 0 0 0 3
## 37 2 0 0 0 0
## 38 0 0 0 0 2
## 39 0 0 0 2 0data <- t(wolf_data)On définit aussi quelques quantités qui nous seront utiles par la suite.
nh <- dim(wolf_data)[1] # nb individus
k <- dim(wolf_data)[2] # nb occ de capture
km1 <- k - 1 # nb occ de recapture
eff <- rep(1, nh) # nb d'individus avec cette histoire particulièreOn récupère l’occasion de première capture, ainsi que l’état de première capture.
fc <- NULL
init.state <- NULL
for (i in 1:nh){
temp <- 1:k
fc <- c(fc, min(which(wolf_data[i,] != 0)))
init.state <- c(init.state, wolf_data[i, fc[i]])
}On source la fonction qui calcule la déviance du modèle.
source('code/dev_phispdelta_age.R')On définit des valeurs initiales par les paramètres.
binit <- runif(5)Et on peut lancer la minimisation de la déviance.
tmpmin <- optim(par = binit,
fn = dev_phispdelta_age,
gr = NULL,
hessian = TRUE,
method = "BFGS",
control = list(trace = 1, REPORT = 1),
data = data,
eff = eff,
e = fc,
garb = init.state,
nh = nh,
km1 = km1)## initial value 190.410103
## iter 2 value 178.974548
## iter 3 value 177.510691
## iter 4 value 176.784391
## iter 5 value 176.121648
## iter 6 value 175.783981
## iter 7 value 175.573982
## iter 8 value 175.463561
## iter 9 value 175.456154
## iter 10 value 175.456037
## iter 11 value 175.456033
## iter 11 value 175.456033
## iter 11 value 175.456033
## final value 175.456033
## convergedOn récupère les estimations sur l’échelle \([0, 1]\).
x <- plogis(tmpmin$par)On calcule aussi les intervalles de confiance.
SElogit <- sqrt(diag(solve(tmpmin$hessian)))
lb <- plogis(tmpmin$par - 1.96 * SElogit)
ub <- plogis(tmpmin$par + 1.96 * SElogit)Les paramètres estimés sont les suivants.
nom_param <- c("Prop initiale état loups",
"Prob de survie des loups",
"Prob de survie des hybrides",
"Prob de détection",
"Prob d'assignation")
est <- data.frame(round(x,2), paste0(round(lb,2), "-", round(ub,2)))
colnames(est) <- c("max-vrais", "int-de-confiance")
row.names(est) <- nom_param
est## max-vrais int-de-confiance
## Prop initiale état loups 0.31 0.21-0.43
## Prob de survie des loups 0.63 0.39-0.82
## Prob de survie des hybrides 0.81 0.59-0.93
## Prob de détection 0.46 0.31-0.61
## Prob d'assignation 0.85 0.75-0.91Estimation de la prévalence
On charge le package HMM qui va nous permettre d’implémenter l’algorithme de Viterbi.
library(HMM)On source la fonction qui applique Viterbi et calcule la prévalence.
source('code/viterbi_phispdelta.R')On calcule la prévalence par occasion de capture. La prévalence donnée ici est celle d’hybrides. Le complémentaire donne la prévalence de loups.
prev_obs <- viterbi_phispdelta(data = wolf_data,
fc = fc,
k = k,
pi = x[1],
phi1 = x[2],
phi2 = x[3],
p = x[4],
delta = x[5],
n.states = 3)
1 - prev_obs## [1] 0.2727273 0.3333333 0.2000000 0.2000000 0.1818182On peut prendre en compte l’incertude sur l’estimation des paramètres du modèle de capture-recapture. Pour ce faire, on utilise un bootstrap non-paramétrique.
On définit d’abord le nombre d’échantillons boostrap.
nb_bootstrap <- 100Puis on rééchantillonne dans les histoires de capture, avec remise, on estime les paramètres et on applique Viterbi et le calcul de la prévalence.
phispipdelta_pseudo <- matrix(NA, nrow = nb_bootstrap, ncol = k)
for (jj in 1:nb_bootstrap){
# rééchantillonne dans les histoires de capture
pseudo_ind <- sample(nrow(wolf_data), replace = T)
pseudo <- wolf_data[pseudo_ind, 1:k]
# ajuste le modèle
data <- t(pseudo)
fc <- NULL
init.state <- NULL
for (kk in 1:nrow(pseudo)){
temp <- 1:ncol(pseudo)
fc <- c(fc,min(which(pseudo[kk,]!=0)))
init.state <- c(init.state,pseudo[kk,fc[kk]])
}
binit <- runif(5)
tmpmin <- optim(par = binit,
fn = dev_phispdelta_age,
gr = NULL,
hessian = TRUE,
method = "BFGS",
control = list(trace = 0),
data = data,
eff = eff,
e = fc,
garb = init.state,
nh = nh,
km1 = km1)
x <- plogis(tmpmin$par)
# applique Viterbi et calcule la prévalence
phispipdelta_pseudo[jj,] <- viterbi_phispdelta(
data = pseudo,
fc = fc,
k = k,
pi = x[1],
phi1 = x[2],
phi2 = x[3],
p = x[4],
delta = x[5],
n.states = 3)
}On calcule un intervalle de confiance pour la prévalence d’hybrides.
res <- rbind(1-prev_obs,
apply(1-phispipdelta_pseudo, 2, quantile, probs = c(2.5, 97.5)/100))
colnames(res) <- paste0("Occ capture ", 1:k)
row.names(res)[1] <- "prév estimée"
round(t(res), 2)## prév estimée 2.5% 97.5%
## Occ capture 1 0.27 0.09 0.61
## Occ capture 2 0.33 0.10 0.61
## Occ capture 3 0.20 0.00 0.50
## Occ capture 4 0.20 0.00 0.50
## Occ capture 5 0.18 0.00 0.50Cas d’étude distribution : estimation de l’hétérogénéité de détection d’une espèce
Ajustement du modèle
On commence par lire les données.
wolf_data <- read_csv("dat/wolf_occupancy.csv")
head(wolf_data)## # A tibble: 6 x 10
## idsite occ1 occ2 occ3 occ4 effort altitude foret X Y
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 8 0 0 0 0 -0.784 -1.03 -1.25 585000 6115000
## 2 9 0 0 0 0 -0.784 1.51 -1.25 595000 6115000
## 3 10 0 0 0 0 -0.784 0.808 -1.25 605000 6115000
## 4 11 0 0 0 0 -0.743 0.675 -1.25 615000 6115000
## 5 12 0 0 0 0 -0.703 0.683 -1.25 625000 6115000
## 6 13 0 0 0 0 -0.703 0.507 -1.25 635000 6115000On ne prend que les histoires de capture des sites, et on regroupe par histoire unique, ce qui permet de réduire drastiquement les temps de calcul.
pooled_dat <- plyr::count(wolf_data[, 2:5], vars = c("occ1","occ2","occ3","occ4"))
dat <- pooled_dat[,1:4]
eff <- pooled_dat[,5]
nh <- nrow(dat)
k <- ncol(dat)
tdat <- t(dat) # transposeOn source la fonction qui calcule la déviance du modèle avec faux positifs et hétérogénéité de détection.
source("code/dev_occufphet.R")On suspect des minima locaux dans la déviance du modèle. On répète la minimisation plusieurs fois, en partant de valeurs initiales différentes à chaque fois.
set.seed(1979)
n.repet <- 100
inits <- matrix(NA, nrow = n.repet, ncol = 7)
mle <- matrix(NA, nrow = n.repet, ncol = 7)
dev <- rep(NA, nrow = n.repet)
for (i in 1:n.repet){
binit <- runif(7)
tmpmin <- optim(par = binit,
fn = dev_occufphet,
gr = NULL,
hessian = FALSE,
method = "BFGS",
control = list(trace = 0),
data = tdat,
eff = eff,
nh = nh,
k = k)
inits[i,] <- binit
mle[i,] <- plogis(tmpmin$par)
dev[i] <- tmpmin$value
}On jette un coup d’oeil aux déviances obtenues.
dev %>%
as_tibble() %>%
ggplot() +
aes(x = value) +
geom_histogram()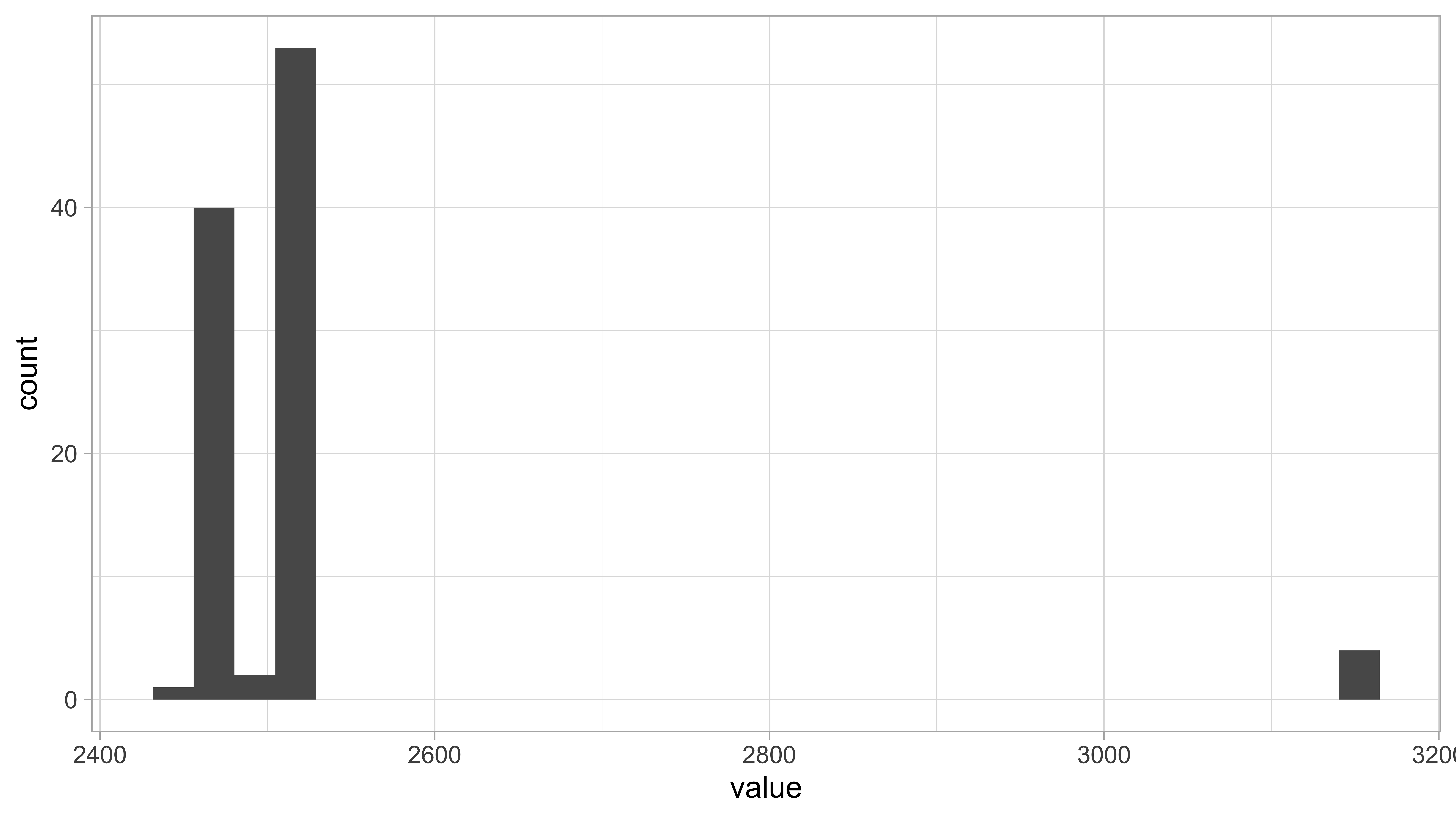
On sélectionne la déviance la plus petite, et on récupère les estimations correspondant sur l’échelle \([0, 1]\).
index <- which(dev == min(dev))
mle[index,]## [1] 6.098218e-02 4.879364e-02 4.814402e-02 1.000000e+00 1.115610e-41
## [6] 3.936977e-01 9.329134e-01On calcule aussi les intervalles de confiance. On refait la minimisation avec les valeurs initiales qui ont conduit au minimum global de la déviance, et en calculant la hessienne (qu’on n’avait pas calculé au-dessus pour accélérer l’étape de minimisation).
binit <- inits[index,]
tmpmin <- optim(par = binit,
fn = dev_occufphet,
gr = NULL,
hessian = TRUE,
method = "BFGS",
control = list(trace = 0),
data = tdat,
eff = eff,
nh = nh,
k = k)
x <- plogis(tmpmin$par)
SElogit <- sqrt(diag(matlib::Ginv(tmpmin$hessian)))
lb <- plogis(tmpmin$par - 1.96 * SElogit)
ub <- plogis(tmpmin$par + 1.96 * SElogit)Les paramètres estimés sont les suivants.
nom_param <- c("Prop of sites in class A",
"Prob occupancy",
"Prob false+ detection in sites A",
"Prob true+ detection in sites A",
"Prob false+ detection in sites B",
"Prob true+ detection in sites B",
"Prob classify a true+ detection as unambiguous")
est <- data.frame(round(x,2), paste0(round(lb,2), "-", round(ub,2)))
colnames(est) <- c("max-vrais", "int-de-confiance")
row.names(est) <- nom_param
est## max-vrais int-de-confiance
## Prop of sites in class A 0.06 0.04-0.09
## Prob occupancy 0.05 0.04-0.06
## Prob false+ detection in sites A 0.05 0.03-0.08
## Prob true+ detection in sites A 1.00 1-1
## Prob false+ detection in sites B 0.00 0-0
## Prob true+ detection in sites B 0.39 0.35-0.43
## Prob classify a true+ detection as unambiguous 0.93 0.9-0.95Visualiser l’hétérogénéité
On charge le package HMM qui va nous permettre d’implémenter l’algorithme de Viterbi.
library(HMM)On définit les quantités nécessaires.
pi <- est[1,1]
psi1 <- est[2,1]
pA10 <- est[3,1]
pA11 <- est[4,1]
pB10 <- est[5,1]
pB11 <- est[6,1]
delta <- est[7,1]
# init-state prob
PI1 <- c(1 - pi, pi)
PI2 <- matrix(c(1 - psi1, 0, psi1, 0, 0, 1 - psi1, 0, psi1),
nrow = 2,
ncol = 4,
byrow = T)
PI <- PI1 %*% PI2 # sum(PI)=1!
# transition matrix
gamA <- gamB <- epsA <- epsB <- 0
A <- matrix(c(1 - gamA, 0, gamA, 0,
0, 1 - gamB, 0, gamB,
epsA, 0, 1 - epsA, 0,
0, epsB, 0, 1 - epsB),
nrow = 4,
ncol = 4,
byrow = T)
# obs matrix
B1 <- matrix(c(1 - pA10, 0, pA10,
1 - pB10, 0, pB10,
1 - pA11, pA11, 0,
1 - pB11, pB11, 0),
nrow = 4,
ncol = 3,
byrow = T)
B2 <- matrix(c(1, 0, 0,
0, delta, 1 - delta,
0, 0, 1),
nrow = 3,
ncol = 3,
byrow = T)
B <- B1 %*% B2On construit le modèle de Markov caché.
hmm <- initHMM(
States = c("NOA", "NOB", "OA", "OB"), # states non-occ A, non-occ B, occ A, occ B
Symbols = c("0", "1", "2"), # 0 = non-detected, 1 = seen and assigned certain, 2 = seen and assigned uncertain
startProbs = PI, # initial states
transProbs = A,
emissionProbs = B)
print(hmm)## $States
## [1] "NOA" "NOB" "OA" "OB"
##
## $Symbols
## [1] "0" "1" "2"
##
## $startProbs
## NOA NOB OA OB
## 0.893 0.057 0.047 0.003
##
## $transProbs
## to
## from NOA NOB OA OB
## NOA 1 0 0 0
## NOB 0 1 0 0
## OA 0 0 1 0
## OB 0 0 0 1
##
## $emissionProbs
## symbols
## states 0 1 2
## NOA 0.95 0.0000 0.0500
## NOB 1.00 0.0000 0.0000
## OA 0.00 0.9300 0.0700
## OB 0.61 0.3627 0.0273On applique Viterbi.
viterbi_res <- matrix(NA, nrow(dat), ncol(dat))
for (i in 1:nrow(dat)){
current_encounter_history <- dat[i,]
# calculate Viterbi path
current_obs <- as.character(current_encounter_history)
viterbi_res[i,] <- viterbi(hmm, current_obs)
}
viterbi_res # chaque site est dans un tat et un seul## [,1] [,2] [,3] [,4]
## [1,] "NOA" "NOA" "NOA" "NOA"
## [2,] "OB" "OB" "OB" "OB"
## [3,] "NOA" "NOA" "NOA" "NOA"
## [4,] "OB" "OB" "OB" "OB"
## [5,] "OB" "OB" "OB" "OB"
## [6,] "NOA" "NOA" "NOA" "NOA"
## [7,] "OB" "OB" "OB" "OB"
## [8,] "OB" "OB" "OB" "OB"
## [9,] "OB" "OB" "OB" "OB"
## [10,] "OB" "OB" "OB" "OB"
## [11,] "OB" "OB" "OB" "OB"
## [12,] "OB" "OB" "OB" "OB"
## [13,] "OB" "OB" "OB" "OB"
## [14,] "OB" "OB" "OB" "OB"
## [15,] "NOA" "NOA" "NOA" "NOA"
## [16,] "OB" "OB" "OB" "OB"
## [17,] "OB" "OB" "OB" "OB"
## [18,] "OB" "OB" "OB" "OB"
## [19,] "OB" "OB" "OB" "OB"
## [20,] "OB" "OB" "OB" "OB"
## [21,] "OB" "OB" "OB" "OB"
## [22,] "OB" "OB" "OB" "OB"
## [23,] "OB" "OB" "OB" "OB"
## [24,] "OB" "OB" "OB" "OB"
## [25,] "OA" "OA" "OA" "OA"
## [26,] "OB" "OB" "OB" "OB"
## [27,] "OB" "OB" "OB" "OB"
## [28,] "NOA" "NOA" "NOA" "NOA"
## [29,] "OB" "OB" "OB" "OB"
## [30,] "NOA" "NOA" "NOA" "NOA"
## [31,] "NOA" "NOA" "NOA" "NOA"
## [32,] "OB" "OB" "OB" "OB"
## [33,] "OB" "OB" "OB" "OB"On peut comparer les observations aux états reconstitués.
cbind(viterbi_res, dat)## 1 2 3 4 occ1 occ2 occ3 occ4
## 1 NOA NOA NOA NOA 0 0 0 0
## 2 OB OB OB OB 0 0 0 1
## 3 NOA NOA NOA NOA 0 0 0 2
## 4 OB OB OB OB 0 0 1 0
## 5 OB OB OB OB 0 0 1 1
## 6 NOA NOA NOA NOA 0 0 2 0
## 7 OB OB OB OB 0 1 0 0
## 8 OB OB OB OB 0 1 0 1
## 9 OB OB OB OB 0 1 0 2
## 10 OB OB OB OB 0 1 1 0
## 11 OB OB OB OB 0 1 1 1
## 12 OB OB OB OB 0 1 1 2
## 13 OB OB OB OB 0 1 2 0
## 14 OB OB OB OB 0 1 2 1
## 15 NOA NOA NOA NOA 0 2 0 0
## 16 OB OB OB OB 0 2 1 2
## 17 OB OB OB OB 1 0 0 0
## 18 OB OB OB OB 1 0 0 2
## 19 OB OB OB OB 1 0 1 0
## 20 OB OB OB OB 1 0 1 1
## 21 OB OB OB OB 1 0 2 0
## 22 OB OB OB OB 1 1 0 0
## 23 OB OB OB OB 1 1 0 1
## 24 OB OB OB OB 1 1 1 0
## 25 OA OA OA OA 1 1 1 1
## 26 OB OB OB OB 1 1 2 0
## 27 OB OB OB OB 1 2 0 1
## 28 NOA NOA NOA NOA 2 0 0 0
## 29 OB OB OB OB 2 0 0 1
## 30 NOA NOA NOA NOA 2 0 0 2
## 31 NOA NOA NOA NOA 2 0 2 0
## 32 OB OB OB OB 2 1 0 0
## 33 OB OB OB OB 2 1 0 2Pour chaque site, on cherche si c’est un classe A ou un classe B.
class_site <- NULL
for (i in 1:nrow(dat)){
if (unique(grepl('A', viterbi_res[i,])) == TRUE) temp <- 'A'
if (unique(grepl('B', viterbi_res[i,])) == TRUE) temp <- 'B'
class_site = c(class_site,temp)
}
class_site## [1] "A" "B" "A" "B" "B" "A" "B" "B" "B" "B" "B" "B" "B" "B" "A" "B" "B" "B" "B"
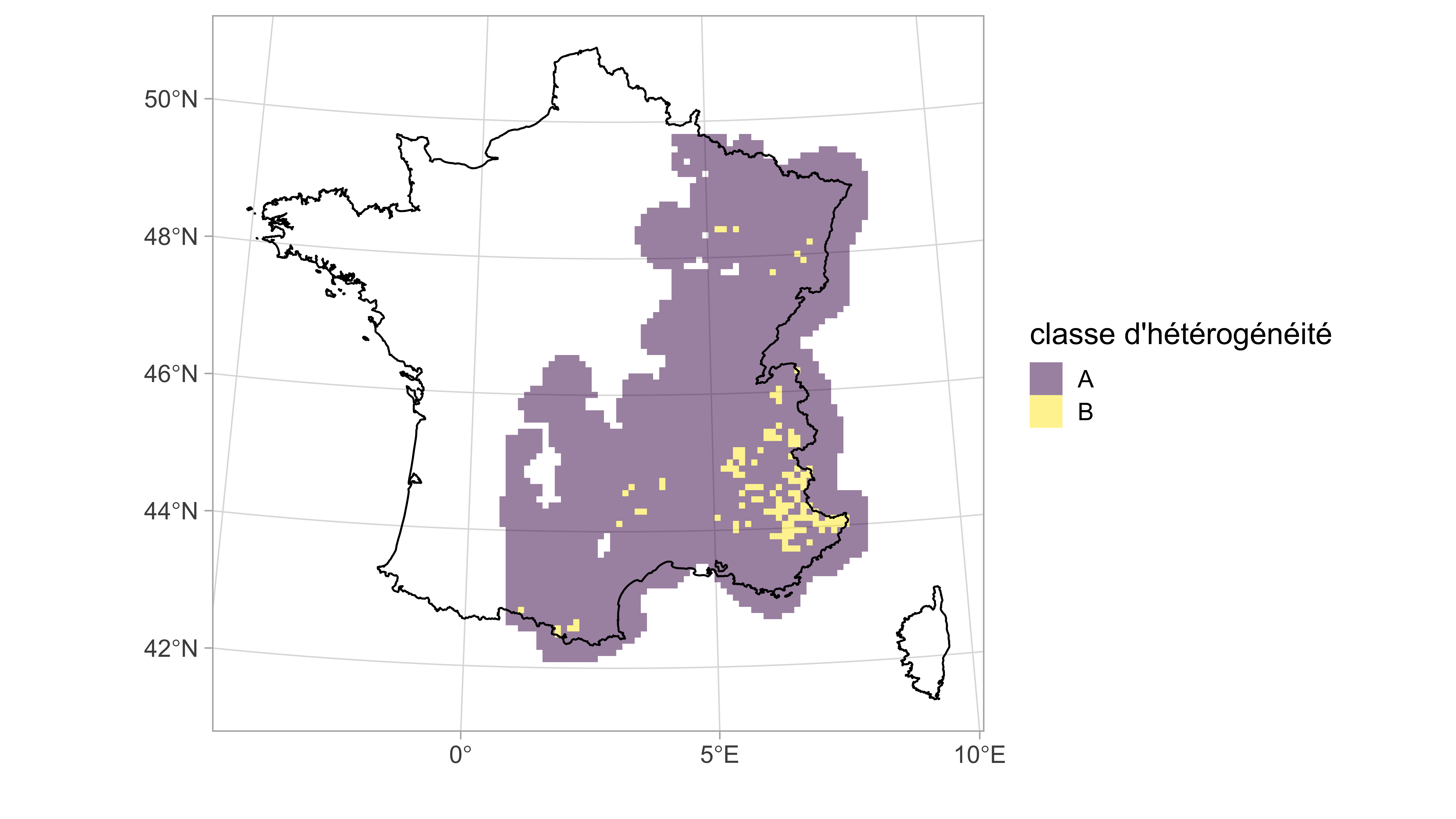
## [20] "B" "B" "B" "B" "B" "A" "B" "B" "A" "B" "A" "A" "B" "B"On peut alors faire une carte avec les sites de type A et ceux de type B. Pour ce faire, il faut associer un état à tous les sites.
all_sites <- wolf_data[, 2:5]
viterbi_res <- matrix(NA, nrow(all_sites), ncol(all_sites))
for (i in 1:nrow(all_sites)){
current_encounter_history <- all_sites[i,]
# calculate Viterbi path
current_obs <- as.character(current_encounter_history)
viterbi_res[i,] <- viterbi(hmm, current_obs)
}
class_site <- NULL
for (i in 1:nrow(all_sites)){
if (unique(grepl('A', viterbi_res[i,])) == TRUE) temp <- 'A'
if (unique(grepl('B', viterbi_res[i,])) == TRUE) temp <- 'B'
class_site <- c(class_site,temp)
}On peut calculer le nombre de sites A et B.
table(class_site)## class_site
## A B
## 3090 121On crée un raster à partir des classes de sites.
class_site2 <- data.frame('class' = class_site,
'x' = wolf_data$X,
'y' = wolf_data$Y) %>%
mutate(class = if_else(class == "A", 1, 2),
x = as.numeric(x),
y = as.numeric(y))
library(raster)
rasterOptions("ascii")
raster_class <- rasterFromXYZ(cbind(class_site2[,'x'],
class_site2[,'y'],
class_site2[,'class']))Puis on visualise.
# font de carte france
france <- st_read("dat/france_union_departements.shp")## Reading layer `france_union_departements' from data source `/Users/oliviergimenez/Dropbox/OG/GITHUB/code_livre_variables_cachees/dat/france_union_departements.shp' using driver `ESRI Shapefile'
## Simple feature collection with 1 feature and 12 fields
## geometry type: MULTIPOLYGON
## dimension: XY
## bbox: xmin: 99217.1 ymin: 6049646 xmax: 1242417 ymax: 7110480
## projected CRS: RGF93_Lambert_93st_crs(france) <- 2154 # Lambert 93
# convertit en SpatialPointsDataFrame
class_pts <- rasterToPoints(raster_class, spatial = TRUE)
class_pts$layer <- if_else(class_pts$layer == 1, "A", "B")
# convertit en dataframe
class_df <- data.frame(class_pts)
ggplot() +
geom_raster(data = class_df , aes(x = x, y = y, fill = as_factor(layer))) +
geom_sf(data = france %>% st_boundary()) +
labs(fill = "classe d'hétérogénéité") +
scale_fill_viridis_d(alpha = 0.5) +
labs(x = "",
y = "")